Regla de Sturges
En estadística descriptiva la Regla de Sturges es un criterio muy utilizado cuando se quiere realizar un histograma de frecuencias ya que con esta regla se calcula el número de clases (o intervalos) necesarios para representar fielmente los datos.
La Regla de Sturges nos propone que dadas N observaciones, el número k de intervalos viene dado por
\[\begin{equation} k = 1 + log_{2}(N) \end{equation}\]Donde N es el número de muestras observadas y k el número óptimo de clases o intervalos.
Hoy en día, con el uso del ordenador, esta regla se hace menos conocida ya que son los propios paquetes estadísticos los que se encargan de calcular el número de intervalos óptimo. De hecho, en R, el comando hist tiene el parámetro breaks = “Sturges” por defecto, aunque siempre podemos modificar este parámetro.
Ahora bien, ¿de donde viene la regla de Sturges?
Sturges consideró un histograma de frecuencias ideal con k intervalos, donde el i-ésimo intervalo contiene un número de muestras dado por el Coeficiente Binomial:
\[\begin{equation} {\displaystyle C_{(k-1,i)}},\, C^{k-1}_i,\, {\displaystyle {k-1 \choose i}={\frac {(k-1)!}{i!(k-1-i)!}}} \end{equation}\]En R se puede calcular el Coeficiente Binomial mediante la función combn(k-1, i).
Por el Teorema Central del Límite sabemos que cuando k aumente el histograma de frecuencias se aproximará a la distribución Normal, por lo que podemos calcular el número de muestras de todos los intervalos, ya que
\[\begin{equation} N = \sum_{i=0}^{k-1}{k-1 \choose i} = (1+1)^{k-1} = 2^{k-1} \end{equation}\]Si aplicamos logaritmos a ambas partes de la ecuación, tenemos:
\[\begin{equation} log_{2}(N) = k - 1 \end{equation}\]por lo que el número óptimo de intervalo k vendrá dado por:
\[\begin{equation} k = 1 + log_{2}(N) \end{equation}\]que es la Regla de Sturges.
Veamos algunos ejemplos donde podamos ver el ajuste dado por este criterio. Vamos a considerar 500 muestras provenientes de una distribución normal de media 10 y desviación típica 2.
x <- rnorm(500, mean = 10, sd = 2)Ahora realizamos tres histogramas con estos datos em los que vamos a aplicar la Regla de Sturges y una aproximación por debajo y por encima del número de intervalos propuestos por la Regla de Sturges. Si calculamos el número de intervalos porpuestos por la Regla de Sturges vemos que:
\[\begin{equation} k = 1 + log_{2}(N) = 1 + log_{2}(500) \approx 10 \end{equation}\]Es decir, 10 intervalos.
par(mfrow = c(1, 3))
hist(x, breaks = 5, col = "lightblue", main = "breaks = 5")
hist(x, breaks = "Sturges", col = "lightblue", main = "breaks = Sturges")
hist(x, breaks = 20, col = "lightblue", main = "breaks = 20")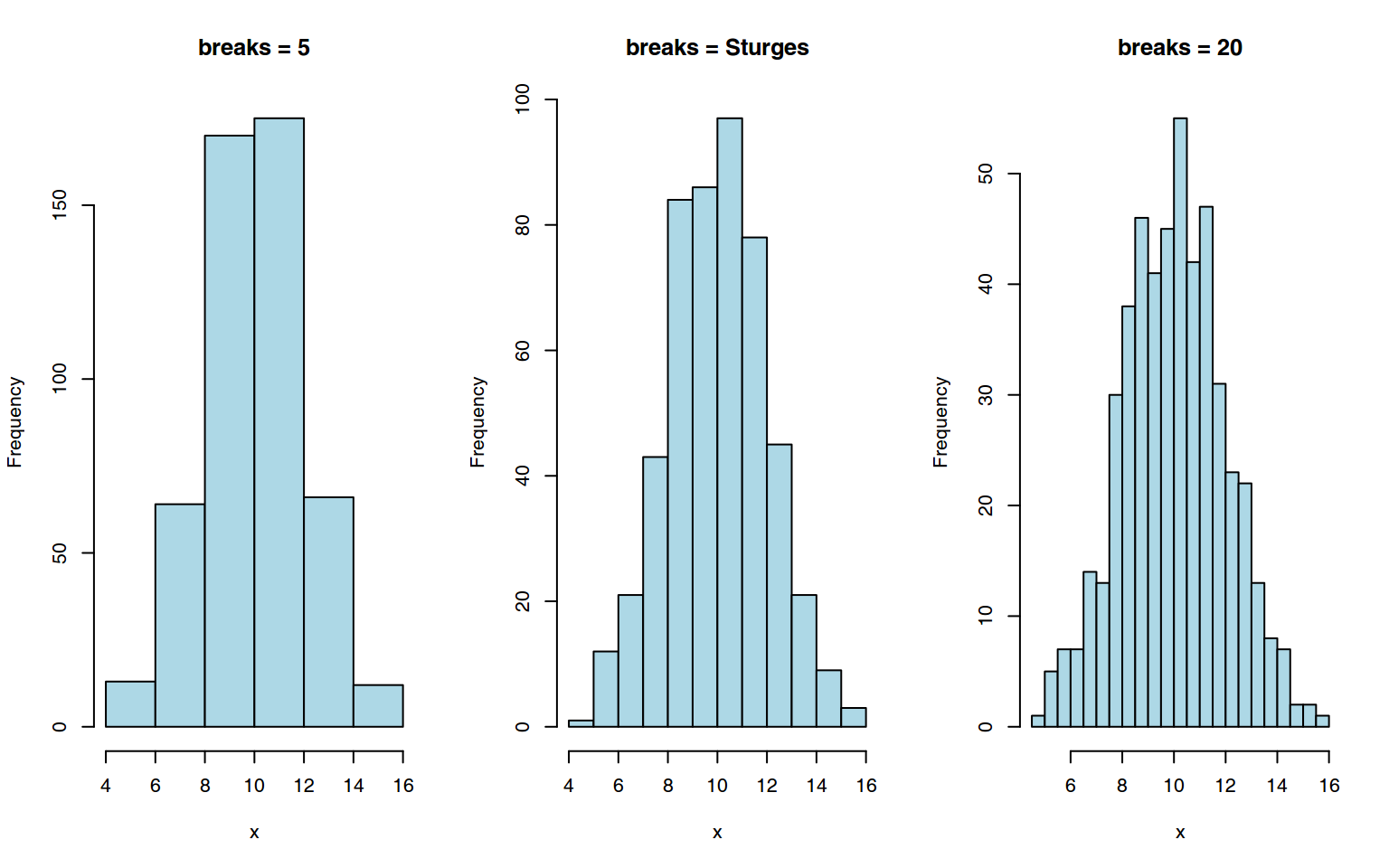
FIGURA 1: Tres aproximaciones a la distribución de las 500 observaciones siguiendo tres criterios.
Observamos que al usar la regla Sturges no obtenemos exactamente 10 clases, ya que la función intenta ajustar el número de intervalos óptimo con unos cortes naturales. En esta caso, los cortes coinciden con valores enteros.
Bibliografía
Altman, Naomi, and Martin Krzywinski. 2015. “Simple Linear Regression.” Nature Methods 12 (11): 999–1000.
———. 2016a. “Points of Significance: Analyzing Outliers: Influential or Nuisance?” Nature Methods 13 (4): 281–82.
———. 2016b. “Points of Significance: Regression Diagnostics.” Nature Methods 13 (5): 385–86.
Armitage, Emily G., and Coral Barbas. 2014. “Metabolomics in Cancer Biomarker Discovery: Current Trends and Future Perspectives.” J Pharm Biomed Anal 87 (January). Centre for Metabolomics; Bioanalysis (CEMBIO), Faculty of Pharmacy, Universidad San Pablo CEU, Campus Monteprincipe, Boadilla del Monte, 28668 Madrid, Spain.: 1–11. doi:10.1016/j.jpba.2013.08.041.
Čuperlović-Culf, Miroslava. 2013. “5 - Metabolomics Data Analysis – Processing and Analysis of a Dataset.” In {NMR} Metabolomics in Cancer Research, edited by Miroslava Čuperlović-Culf, 261–333. Woodhead Publishing Series in Biomedicine. Woodhead Publishing. doi:http://dx.doi.org/10.1533/9781908818263.261.
Fox, J. 1997. Applied Regression Analysis, Linear Models, and Related Methods. SAGE Publications.
Krzywinski, Martin, and Naomi Altman. 2014. “Points of Significance: Two-Factor Designs.” Nature Methods 11 (12): 1187–8.
Kuehl, R.O., and M.G. Osuna. 2001. Diseño de Experimentos: Principios Estadísticos de Diseño Y análisis de Investigación. Matemáticas (Thomson). International Thomson Editores, S. A. de C. V.
Martínez-Arranz, Ibon, Rebeca Mayo, Miriam Pérez-Cormenzana, Itziar Mincholé, Lorena Salazar, Cristina Alonso, and José M. Mato. 2015. “Enhancing Metabolomics Research Through Data Mining.” Journal of Proteomics 127, Part B (0): 275–88. doi:http://dx.doi.org/10.1016/j.jprot.2015.01.019.
Pulido, H.G., R. de la Vara Salazar, P.G. González, C.T. Martínez, and M.C.T. Pérez. 2004. Análisis Y Diseño de Experimentos. McGraw-Hill.
Xie, Yihui. 2014. “Knitr: A Comprehensive Tool for Reproducible Research in R.” In Implementing Reproducible Computational Research, edited by Victoria Stodden, Friedrich Leisch, and Roger D. Peng. Chapman; Hall/CRC. http://www.crcpress.com/product/isbn/9781466561595.
———. 2015. Dynamic Documents with R and Knitr. 2nd ed. Boca Raton, Florida: Chapman; Hall/CRC. http://yihui.name/knitr/.
———. 2016. Knitr: A General-Purpose Package for Dynamic Report Generation in R. http://yihui.name/knitr/.